機能詳細4 受容体構造に基づく分子設計2 -相互作用解析-
タンパク質とリガンドの相互作用を正しく理解することは、化合物を評価する上で非常に重要です。MOEでは、様々な視点からンタパク質とリガンドの結合状態の特徴付けを行うことができます。
タンパク質ーリガンド相互作用解析
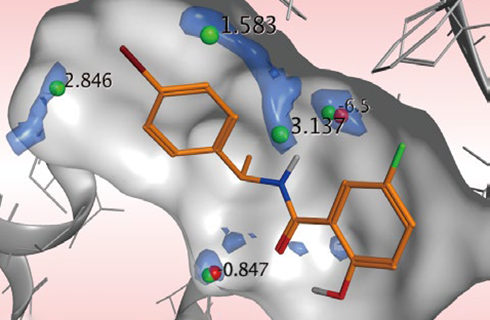
3D-RISM※1法による溶媒解析
統計力学的手法により、高速かつ高精度に溶媒の密度分布や溶媒和エネルギーを求め、タンパク質やリガンドの周囲における溶媒の確率密度分布を可視化します。溶媒和エネルギーに基づく水素結合の評価、水分子によるブリッジの理解、疎水性相互作用など、相互作用に関わる溶媒効果を詳細に検討できます。また、溶媒解析の結果を基にして、水分子の位置を予測できます。予測した各水分子の位置は、自由エネルギー差により評価されます。タンパク質ーリガンド相互作用における重要な水分子を、水素原子の向きも含めて高精度に予測できます。
- ※1Kavalenko, A. ; Hirata, F. Self-Consistent Description of a Metal-Water Interface by the Kohn-Sham Density Functional
Theory and the Three Dimensional Reference Interaction Site Model. J. Chem. Phys. 1999, 110, 10095-10112.
Protein Ligand lnteraction Fingerprints (PLIF)
PLIFは、結合部位のアミノ酸残基とリガンドとの間の相互作用をフィンガープリントに変換し、複数のタンパク質—リガンド間相互作用を比較できます。タンパク質ー低分子間では水素 結合、イオン結合、表面接触、配位結合、アレーン接触に基づき、タンパク質ータンパク質間ではタンパク質表面パッチの接触(疎水ー疎水、電荷ー電荷など)による相互作用に基づき解析します。
PLIFには次のような解析機能があります。
- バーコード表示による相互作用の要約表示
- ヒストグラムによる重要残基表示
- 活性/不活性を区別する相互作用の検出
- 同様の相互作用パターンを持つ化合物の検索
- (タンパク質ータンパク質間)相互作用パターンに基づくクラスタリングとエピトープの予測

結合自由エネルギーの推算
AMBER※2の熱力学的積分法のインターフェースにより、相対的なリガンド受容体問の結合自由エネルギーを簡単に計算できます。
- ※2別途入手が必要
プロジェクトデータ管理
MOE Project
立体構造、配列、アラインメント情報、電子密度マップ、PLIF、物性値等のデータを一つの データベース(プロジェクトデータベース)に集約しSBDDのためのデータ管理を簡易化します。立体構造のデータは、インハウスデータ、PSIL0※3、RCSBから取得でき、立体構造の重ね合わせ、配列のアラインメント、構造/配列アノテーション、分子記述子の算出なども自動で行います。
- ※3PSILO: https://www.molsis.co.jp/lifescience/psilo/
Project Search
プロジェクトデータベースに対して、PDB IDや種などのキーワード、リガンド構造、アミノ酸残基位置、立体構造の類似度、配列相同性などを組み合わせて柔軟な検索ができます。論文の著者名やタイトルによる検索もでき、PubMedや文献へのリンクが可能です。
リガンド結合部位の特徴づけ
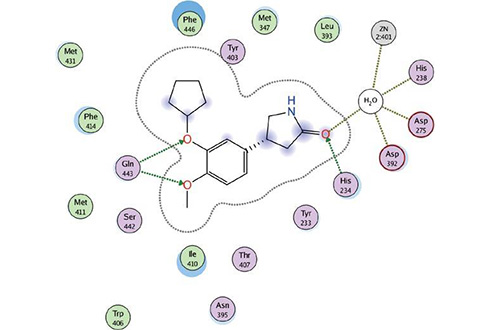
ドッキングサイトの2次元表示
タンパク質リガンド複合体からリガンド結合部位の2次元図を表示します。周辺残基の位置関係や相互作用、溶媒露出度が直感的に分かるように表示されます。水素結合、イオン結合、π-π相互作用、CH-π 相互作用、カチオン-π相互作用、ハロゲン結合、配位結合などの相互作用を、それぞれ色分けして表示します。相互作用は、MOEウィンドウの3D表示と対応しています。
タンパク質表面解析
分子の溶媒露出表面や相互作用表面を表示し、分子表面をさまざまな性質で色分けします。原子色、水素結合性、静電ポテンシャル、温度因子、親水性/疎水性等での色付けや部分毎に任意の着色もできます。タンパク質ータンパク質間相互作用やタンパク質の凝集に関与する疎水性/正電荷/負電荷の特性を持つ分子表面を抽出できます。
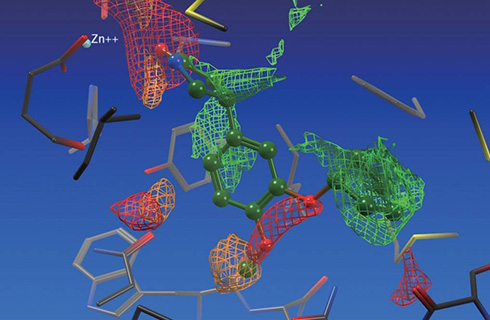
分子表面付近の特性解析
物理化学的性質や知識ベースの情報に基づいて、表面付近の空問において特定の性質を持つ原子が存在する可能性が高い領域を表示します。
- Contact Preference
PDBに対する統計解析データを基に、親水性/疎水性原子が存在する可能性が高い領域を表示 - Electrostatic Map
Poisson-Boltzmann静電ポテンシャル計算を基に、水素結合供与体や受容体、疎水性原子のポテンシャルの等値面を表示 - Interaction Potential
力場計算を基に、34種の原子タイプのポテンシャルエネルギーの等値面を表示
ポケット探索
リガンド原子の存在可能な位置にアルファ球と呼ばれる小球を配置し、各ポケットの大きさや形状、親水性/疎水性領域の分布を表示します。探索されたポケットは、リガンド結合傾向のスコアであるPLBインデックス※4により評価され、リガンド結合部位を高精度に予測できます。
- ※4Soga, S. ; Shirai, H. ; Kobori, M. ; Hirayama, N. Use of Amino Acid Composition to Predict Ligand-Binding Sites. J. Chem. Inf. Model. 2007, 47, 400-406.
統合計算化学システムMOE に関するお問い合わせ
-
Webでのお問い合わせ
入力フォーム当社はセキュリティ保護の観点からSSL技術を使用しております。
-
お電話でのお問い合わせ
富士通コンタクトライン(総合窓口)
0120-933-200(通話無料)受付時間:9時~12時および13時~17時30分(土曜・日曜・祝日・当社指定の休業日を除く)
