機能詳細2 タンパク質・抗体モデリングと構造解析
アミノ酸配列を入力としてホモロジー検索、配列アラインメントを経てタンパク質の単量体や多量体、抗体の立体構造予測を行います。既知の立体構造や変異体モデルに対する物性推算、表面解析、相互作用解析を行うことで対象タンパク質の特徴や重要残基の知見を得ることが可能です。
タンパク質モデリング
ホモロジー検索
独自のファミリーデータベースを使用し、類似配列を持つタンパク質をドメインごとに検索します。検索結果から取得した立体構造を、構造保持領域の解析やホモロジーモデリングに利用できます。
アミノ酸配列アラインメント
Needleman-Wunsch法に基づいたアルゴリズムによる配列アラインメントを行います。タンパク質のモチーフや立体構造の重なりを考慮したアラインメントの補正機能も搭載しています。
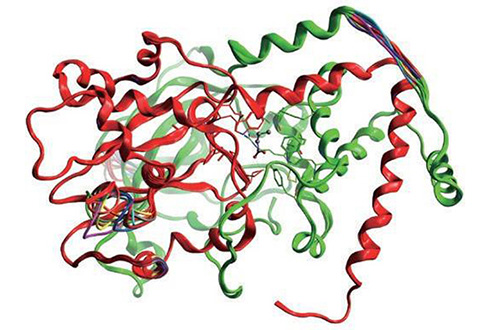
ホモロジーモデリング
- 単量体・多量体モデリング
- 立体構造が既知の鋳型タンパク質に対して、ターゲットアミノ酸配列を当てはめるように立体構造を構築します。鋳型タンパク質を参照できない部分的な主鎖構造は、セグメントマッチ法※1により他の立体構造データを割り当て、側鎖構造は最適なパッキングとなるものをライブラリーから選択します。
複数のタンパク質をテンプレートとすることで、多量体モデルも構築できます。
- ※1Levitt, M. Accurate modeling of protein conformation by automatic segment matching. J. Mol. Biol.1992, 226(2), 507-533.
- 抗体モデリング
- 抗体の相補性決定領域(CDR)やフレームワーク領域に対してモデリングに適したテンプレートを自動・手動選択で決定し、目的の抗体モデルを構築します。Fv 、Fab 、F(ab')、rlg、lg領域など、構築範囲の指定ができます。重鎖あるいは軽鎖のみで構成する単ードメイン抗体(VHH、VLL)や2種類の重鎖と2種類の軽鎖で構成するバイスペシフィック抗体、抗体のフレームワークをヒト由来の配列で置き換えたヒト化抗体も構築可能です。
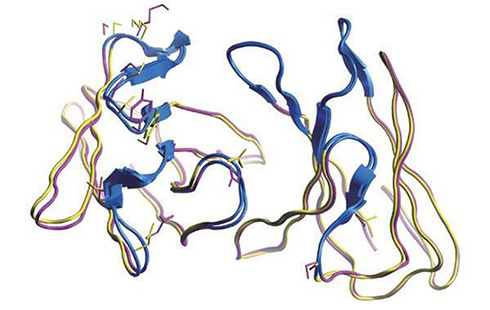
タンパク質ータンパク質ドッキング
タンパク質の各アミノ酸残基を1~3個のビーズに変換した粗視化モデルを使用して、タンパク質の複合体構造を高速に予測します。ドッキング部位の指定も行えます。抗体—抗原ドッキングでは自動的にCDRを検出し、その部位に限定したドッキングが可能です。
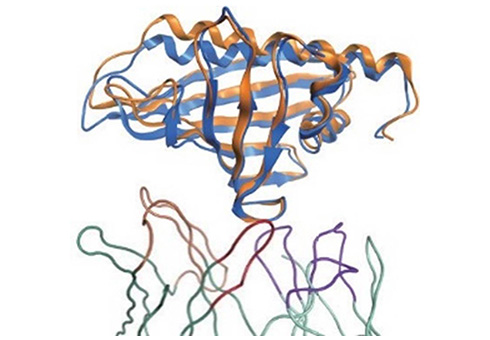
タンパク質デザイン・抗体医薬
タンパク質デザイン・抗体医薬
タンパク質工学/ペプチドデザインにおける任意の変異を導入した際の影習をシミュレーションします。アラニンスキャニング、システインスキャニング(ジスルフィド結合可能位置の探索)なども容易に実行できます。非天然のアミノ酸を用いた変異体の構築もできます。
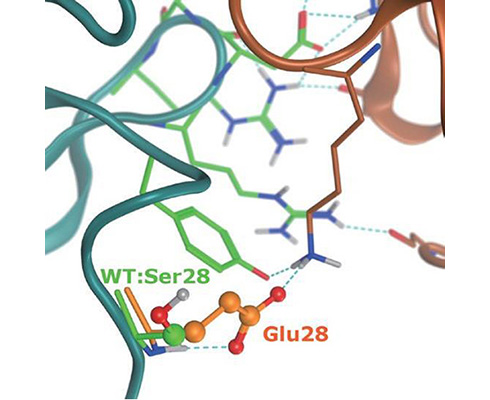
ループグラフティング
任意の部分構造を他の構造より移植した、タンパク質モデルを容易に構築できます。移植による構造のひずみは自動的に緩和されます。
ループ/リンカーモデリング
経験的探索法、またはde novo探索法により任意の残基長のループ/リンカー構造を構築します。リンカー導入後の各ドメインの最適な配置を、剛体の構造最適化により予測することも可能です。
翻訳後修飾モデリング
グリコシル化、アセチル化、メチル化、ニトロシル化、硫酸化、リン酸化などの候補部位を検出し、自動的にモデル構築できます。
フォーカストライブラリー
バーチャルファージディスプレイ法により、突然変異体の膨大な探索空間の中から目的の物性(熱安定性、リガンド親和性等)を持つアミノ酸配列候補を推定します。
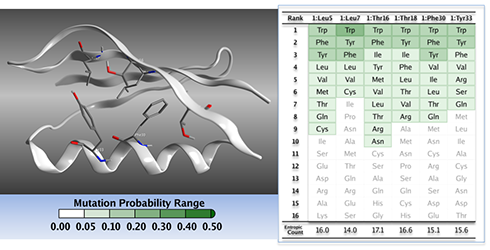
ペプチド・タンパク質構造構築
アミノ酸配列を入力することでペプチドを容易に構築できます 。また、既存の側鎖構造から、点変異体の構築、側鎖配座の探索、主鎖・側鎖の構造最適化、側鎖の再パッキング等、様々な構造探索も行えます。非天然アミノ酸の構築にも対応しています。
エピトープマッピング
タンパク質-タンパク質ドッキングの結果を用いて、抗原の抗体認識部位(エピトープ)などのタンパク質-タンパク質相互作用界面のアミノ酸残基を予測します。水素-重水素交換法などの実験情報を考慮して、相互作用に関与している可能性の高い領域に重み付けした解析も可能です。
タンパク質解析
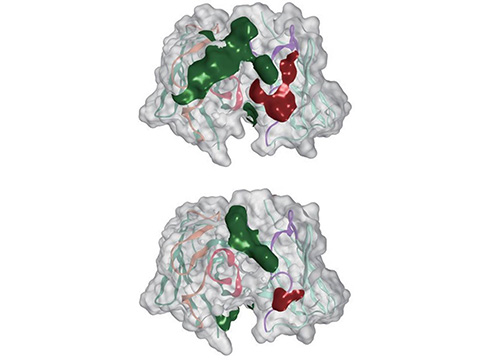
表面パッチ解析
タンパク質表面上の疎水性領域、正電荷領域、負電荷領域を検出、可視化します。さらに各領域を2次元投影し、タンパク質間の表面特性の差異を見やすく描画します。またタンパク質データベースに対して、表面パッチの数や表面積を連続計算できます。
タンパク質物性推算
タンパク質の分子量、疎水性・親水性表面積、ファンデルワールス体積、等電点、総電荷、双極子モーメント、ゼータ電位など96種類の物性をpHと構造変化を考慮して推算できます。アミノ酸残基ごとの物性への寄与も計算可能です。
タンパク質相互作用解析
残基間の水素結合、イオン相互作用、ジスルフィド結合、VDW相互作用を検出します。分子間相互作用や複数分子間に共通する相互作用も検出できます。
構造ひずみチェック
Ramachandranプロットなどの二面角分布や原子間衝突、側鎖配座エネルギーなどを算出し、タンパク質立体構造としての妥当性を評価します。
アミノ酸残基のpKa予測
PROPKA法※2に基づく手法により、タンパク質立体構造から各残基のpKa値を予測します 。
- ※2Li, H. ; Robertson, A. D. ; Jensen, J. H. Very fast empirical prediction and rationalization of protein pKa values.Proteins.2005, 61, 704-721.
ドメインモチーフ検索
タンパク質のドメインを構成する二次構造を表すベクトルの集合をクエリーとして、配列情報に依存しない二次構造の立体配置が類似するタンパク質の検索を行います。
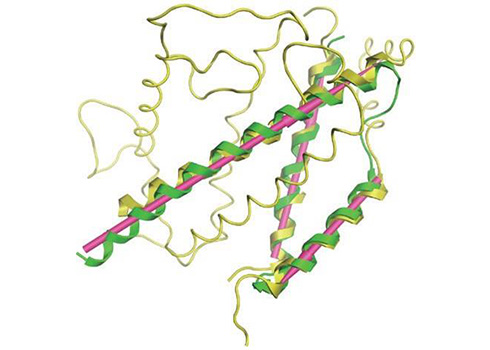
タンパク質重ね合わせ
複数のタンパク質立体構造を、アミノ酸配列、折りたたみ、二次構造の立体配置に基づいて重ね合わせを行います。残基ごとのRMSD値もチェックできます。
統合計算化学システムMOE に関するお問い合わせ
-
Webでのお問い合わせ
入力フォーム当社はセキュリティ保護の観点からSSL技術を使用しております。
-
お電話でのお問い合わせ
富士通コンタクトライン(総合窓口)
0120-933-200(通話無料)受付時間:9時~12時および13時~17時30分(土曜・日曜・祝日・当社指定の休業日を除く)
